Polyadenylatie

Polyadenylatie is een posttranscriptionele modificatie van het eukaryotische pre-mRNA, waarbij door het enzym poly-A-polymerase aan het 3'-eind van het pre-mRNA adenine-nucleotiden worden bevestigd, de poly(A)-staart. Andere posttranscriptionele modificaties van het pre-mRNA zijn onder meer splicing en capping.
De poly(A)-staart wordt niet door het DNA gecodeerd, maar door het enzym poly(A)-polymerase tijdens de productie van het pre-mRNA in de celkern aan het 3'-eind van het pre-mRNA vastgemaakt. Dit mRNA wordt poly(A)+-mRNA genoemd. De lengte van de poly(A)-staart varieert afhankelijk van het organisme. Zo heeft bakkersgist een staart met ongeveer 80 nucleotiden en zoogdieren ongeveer 250 nucleotiden. Aan de poly(A)-staart zit het poly(A)-bindingsproteïne (PABP) dat noodzakelijk is voor het beginnen van de translatie.
Bij de polyadenylatie zijn vele eiwitten betrokken, zoals het eiwit CPSF (cleavage and polyadenylation specificity factor) dat het polyadenylatiesignaal (5'…AAUAAA…3') herkent. Het poly(A)-bindingsproteïne (PABPN1) bindt tijdens de synthese aan de groeiende poly(A)-staart. De poly(A)-staart verkort zich met toenemende ouderdom van het mRNA-molecuul en reguleert zo de halveringstijd van het mRNA-molecuul. In sommige celtypen worden mRNA's met een korte poly(A)-staart opgeslagen in het cytosol voor later gebruik bij de her-polyadenylatie.[1]
Functies
De functies van de poly(A)-staarten zijn nog niet helemaal duidelijk, zeker is echter dat ze zorgen voor een verhoging van de stabiliteit van het mRNA door bescherming tegen afbraak en voor een verhoging van de transleerbaarheid. Een 7-methyl-guanosine-nucleotide (capstructuur) bindt zich aan het 5'-eind en dient eveneens als bescherming tegen afbraak.
Bijna alle eukaryotische mRNA's zijn gepolyadenyleerd[2] met uitzondering van dierlijke replicatie afhankelijke histon mRNA's.[3] Dit zijn de enige mRNAs in eukaryoten die geen poly(A)-staart hebben en in plaats daarvan aan het eind een stam-lusstructuur hebben gevolgd door een purine rijke sequentie, genaamd het histon stroomafwaarts element, dat aangeeft waar het RNA geknipt is en waar het 3'-eind van het histon-mRNA is gevormd.[4]
Vele eukaryotische niet-coderende RNA's worden aan het eind van de transcriptie altijd gepolyadenyleerd. Er zijn echter kleine RNA's waar de poly(A)-staart alleen voorkomt bij tussenvormen en niet bij het volwassen RNA, omdat de einden tijdens de aanmaak worden verwijderd. Dit zijn de microRNA's.[5][6] Maar bij vele lange niet-coderende RNA's is een poly(A)-staart een deel van het volwassen RNA.[7] Lange niet-coderende RNA's vormen een grote groep van regulerende RNA's, zoals het RNA Xist, dat het X-chromosoom inactiveert.
Bij de afbraak van het mRNA is een bacterieel eiwitcomplex, de degradosoom, betrokken.
Alternatieve polyadenylatie
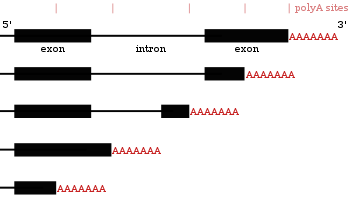
Vele eiwitcoderende genen hebben meer dan één polyadenylatie plek, waardoor een gen voor meerdere mRNA's, die verschillen door hun 3'-eind, kan coderen.[8][9][10] Sinds alternatieve polyadenylatie de lengte van de 3' niet-getransleerde regio verandert, kan het wisselen met bindingsplaatsen voor microRNA's, die een 3' niet-getransleerde regio hebben.[11][12] MicroRNA's neigen naar het onderdrukken van de translatie en naar het bevorderen van de afbraak, ofschoon er voorbeelden zijn van microRNA's die transcripten stabiliseren.[13][14] Alternatieve polyadenylatie kan ook de coderende regio verkorten en zo een mRNA maken voor het coderen van een ander eiwit.[15][16] maar dit komt veel minder voor dan het alleen maar verkorten van de 3' niet-getransleerde regio.[8]
De keuze van de poly(A)-plaats kan beïnvloed worden door buitencellulaire prikkels en hang af van de expressie van de eiwitten die bij de polyadenylatie betrokken zijn.[17][18] De expressie van bijvoorbeeld CstF-64, een ondereenheid van de cleavage stimulatory factor (CstF), verhoogt in macrofagen de reactie oplipopolysacchariden (een groep van bacteriële stoffen die de immuunreactie in gang zetten. Dit zorgt voor het selecteren van zwakke poly(A)-staarten en dus kortere transcripten. Hierdoor worden regulatie-elementen verwijderd in de 3' niet-getranslateerde regionen van mRNA's voor afweer gerelateerde producten als lysozymen en de knip stimulerende TNF-α factor. Deze mRNA's hebben vervolgens een langere halveringstijd en produceren meer van deze eiwitten.[17] Andere RNA-bindingsproteïnen dan die in de polyadenylatie voorkomen en nauw verwant zijn aan het polyadenylatie signaal kunnen ook effect hebben op het gebruik van een polyadenylatie plek.[18][19][20][21][22]
Cytoplasmatische polyadenylatie
Hoewel polyadenylatie meestal plaatsvindt in de celkern komt het ook voor in het cytosol van sommige dierlijke celtypen, namelijk in de kiembaan, gedurende de vroege embryogenese en in post-synaptische plaatsen van zenuwcellen. Dit verlengt de poly(A)-staart van een mRNA met een verkorte poly(A)-staart, zodat het mRNA getranslateerd kan worden.[23][24] Deze verkorte poly(A)-staarten zijn vaak minder dan 20 nucleotiden lang en worden op deze manier verlengd tot 80–150 nucleotiden.[25]
In een vroeg stadium van het muisembryo zorgt cytoplasmatische polyadenylatie van ouderlijke RNA's uit de eicel ervoor dat de cel overleeft en zelfs kan groeien ondanks dat de transcriptie pas begint tijdens de tweede helft van het tweecellig stadium (bij mensen pas in het viercellig stadium).[26][27] In de hersenen is cytoplasmatische polyadenylatie actief tijdens leerprocessen en zou een rol kunnen spelen bij langetermijnpotentiatie, het versterken van de signaaloverdracht van de ene zenuwcel naar de andere door zenuwimpulsen en is belangrijk voor het leren en het onthouden van informatie[28]
Voor cytoplasmatische polyadenylatie zijn de RNA-bindingsproteïnen CPSF en CPEB nodig en mogelijk ook andere RNA-bindingsproteïnen zoals Pumilio.[29] Afhankelijk van het celtype kan de polymerase van hetzelfde type van gepolyadenyleerde polymerase (PAP) zijn dat gebruikt wordt bij de processen in de celkern of het cytoplasmatische polymerase GLD-2.[30]
Geschiedenis
Polyadenylatie werd voor het eerst in 1960 ontdekt als een actief enzym in extracten van celkernen, die ATP in polyadenine konden polymeriseren, maar geen ADP in polyadenine.[31][32] Hoewel polyadenylatie in veel celtypen werd gevonden was de functie tot 1971 onbekend. In dit jaar werden poly(A)-sequenties in mRNA's gevonden.[33][34] In het begin werd gedacht dat polyadenylatie alleen het 3'-eind van het RNA beschermde tegen afbraak door nucleasen. Later werd ontdekt dat polyadenylatie een specifieke rol speelt bij de export uit de kern en bij de translatie. De polymerasen, die verantwoordelijk zijn voor de polyadenylatie, werden voor het eerst gezuiverd en gekenmerkt in de zestiger en zeventiger jaren van de twintigste eeuw. Pas in het begin van de negentiger jaren van de twintigste eeuw werd echter het grote aantal eiwitten ontdekt, dat dit proces controleert.[35]
Zie ook
- ↑ Richter, Joel D. (1999). "Cytoplasmic Polyadenylation in Development and Beyond". Microbiology and Molecular Biology Reviews 63 (2): 446–56. PMC 98972. PMID 10357857
- ↑ Arthur G. Hunt, Ruqiang Xu, Balasubrahmanyam Addepalli, Suryadevara Rao, Kevin P. Forbes, Lisa R. Meeks, Denghui Xing, Min Mo, Hongwei Zhao, 2008, Arabidopsis mRNA polyadenylation machinery: comprehensive analysis of protein-protein interactions and gene expression profiling, BMC Genomics, 9, pp 220, DOI:10.1186/1471-2164-9-220, PMID 18479511, PMC 2391170
- ↑ M. Davila Lopez, T. Samuelsson, 2007, Early evolution of histone mRNA 3' end processing, RNA, 14, 1, pp 1–10, PMID 17998288, DOI:10.1261/rna.782308, PMC 2151031
- ↑ William F. Marzluff, Preetam Gongidi, Keith R. Woods, Maltais Jianping, Lois J., 2002, The Human and Mouse Replication-Dependent Histone Genes, Genomics, 80, 5, pp 487–98, DOI:10.1016/S0888-7543(02)96850-3 PMID 12408966
- ↑ H.K. Saini, S. Griffiths-Jones, A.J. Enright, 2007, Genomic analysis of human microRNA transcripts, Proceedings of the National Academy of Sciences, 104, 45, pp. 17719–24, DOI:10.1073/pnas.0703890104
- ↑ M. Yoshikawa, 2005, A pathway for the biogenesis of trans-acting siRNAs in Arabidopsis, Genes & Development, 19, 18, PMID 16131612, PMC 1221887, pp. 2164–75, DOI:10.1101/gad.1352605
- ↑ Amaral, Paulo P., Mattick, John S. (2008). Noncoding RNA in development. Mammalian Genome 19 (7–8): 454–92. PMID 18839252. DOI: 10.1007/s00335-008-9136-7.
- ↑ a b Y. Shen, G. Ji, B.J. Haas, X. Wu, J. Zheng, G.J. Reese, Q. Li, 2008, Genome level analysis of rice mRNA 3'-end processing signals and alternative polyadenylation , Nucleic Acids Research, 36, 9, pp 3150–61, PMID 18411206, PMC 2396415, DOI:10.1093/nar/gkn158
- ↑ B. Tian, J. Hu, H. Zhang, C.S. Lutz, 2005, A large-scale analysis of mRNA polyadenylation of human and mouse genes, Nucleic Acids Research, 33, 1, pp 201–12, PMID 15647503, DOI:10.1093/nar/gki158, PMC 546146
- ↑ Sven Danckwardt, Matthias W. Hentze, Andreas E. Kulozik, 2008, 3′end mRNA processing: molecular mechanisms and implications for health and disease, The EMBO Journal, 27, 3 , pp 482–98, DOI:10.1038/sj.emboj.7601932, PMID 18256699, PMC 2241648
- ↑ D. Liu, J.M. Brockman, B. Dass, L.N. Hutchins, P. Singh, J.R. McCarrey, C.C. MacDonald, J.H. Graber, 2006, Systematic variation in mRNA 3'-processing signals during mouse spermatogenesis, Nucleic Acids Research, 35, 1, pp 234–46, DOI:10.1093/nar/gkl919, PMID 17158511, PMC 1802579
- ↑ R. Sandberg, J.R. Neilson, A. Sarma, P.A. Sharp, C.B. Burge, 2008, Proliferating Cells Express mRNAs with Shortened 3' Untranslated Regions and Fewer MicroRNA Target Sites, Science, 320, 5883, pp 1643–7, DOI:10.1126/science.1155390, PMID 18566288, PMC 2587246
- ↑ Tili Esmerina, Michaille Jean-Jacques, Calin George Adrian, 2008, Expression and function of micro-RNAs in immune cells during normal or disease state, International Journal of Medical Sciences, 5, 2, pp 73–9, PMID 18392144, PMC 2288788, [1]
- ↑ T. Ghosh, K. Soni, V. Scaria, M. Halimani, C. Bhattacharjee, B. Pillai, 2008, MicroRNA-mediated up-regulation of an alternatively polyadenylated variant of the mouse cytoplasmic -actin gene, Nucleic Acids Research, 36, 19, pp 6318–32, DOI:10.1093/nar/gkn624, PMID 18835850, PMC 2577349
- ↑ F. Alt, A.L. Bothwell, M. Knapp, E. Siden, E. Mather, M. Koshland, D. Baltimore, 1980, Synthesis of secreted and membrane-bound immunoglobulin mu heavy chains is directed by mRNAs that differ at their 3′ends, Cell, 20, 2, pp 293–301, PMID 6771018, DOI:10.1016/0092-8674(80)90615-7
- ↑ B. Tian, Z. Pan, J.Y. Lee, 2007, Widespread mRNA polyadenylation events in introns indicate dynamic interplay between polyadenylation and splicing, Genome Research, 17, 2, pp 156–65, DOI:10.1101/gr.5532707, PMID 17210931, PMC 1781347
- ↑ a b S.A. Shell, C. Hesse, S.M. Morris Jr, C. Milcarek, 2005, Elevated Levels of the 64-kDa Cleavage Stimulatory Factor (CstF-64) in Lipopolysaccharide-stimulated Macrophages Influence Gene Expression and Induce Alternative Poly(A) Site Selection, Journal of Biological Chemistry, 280, 48, pp 39950–61, DOI:10.1074/jbc.M508848200, PMID 16207706
- ↑ a b Sven Danckwardt, Anne-Susan Gantzert, Stephan Macher-Goeppinger, Hans Christian Probst, Marc Gentzel, Matthias Wilm, Hermann-Josef Gröne, Peter Schirmacher, Matthias W. Hentze, 2011, p38 MAPK Controls Prothrombin Expression by Regulated RNA 3′ End Processing, Molecular Cell, 41, 3, pp 298–310, DOI:10.1016/j.molcel.2010.12.032, PMID 21292162
- ↑ Donny D. Licatalosi, Aldo Mele, John J. Fak, Jernej Ule, Melis Kayikci, Sung Wook Chi, Tyson A. Clark, Anthony C. Schweitzer, John E. Blume, 2008, HITS-CLIP yields genome-wide insights into brain alternative RNA processing, Nature, 456, 7221, pp 464–9, DOI:10.1038/nature07488, PMID 18978773, PMC 2597294
- ↑ T. Hall-Pogar, S. Liang, L.K. Hague, C.S. Lutz, 2007, Specific trans-acting proteins interact with auxiliary RNA polyadenylation elements in the COX-2 3'-UTR, RNA, 13, 7, pp 1103–15, DOI:10.1261/rna.577707, PMID 17507659, PMC 1894925
- ↑ Sven Danckwardt, Isabelle Kaufmann, Marc Gentzel, Konrad Foerstner, Anne-Susan Gantzert, Niels H. Gehring, Gabriele Neu-Yilik, Peer Bork, Walter Keller, 2007, Splicing factors stimulate polyadenylation via USEs at non-canonical 3′end formation signals, The EMBO Journal, 26, 11, pp 2658–69, DOI:10.1038/sj.emboj.7601699, PMID 17464285, PMC 1888663
- ↑ A.J. Wood, R. Schulz, K. Woodfine, K. Koltowska, C.V. Beechey, J. Peters, D. Bourc'his, R.J. Oakey, 2008, Regulation of alternative polyadenylation by genomic imprinting, Genes & Development, 22, 9, pp 1141–6, DOI:10.1101/gad.473408
- ↑ Meijer, H. A.; Bushell, M.; Hill, K.; Gant, T. W.; Willis, A. E.; Jones, P.; De Moor, C. H. (2007). "A novel method for poly(A) fractionation reveals a large population of mRNAs with a short poly(A) tail in mammalian cells". Nucleic Acids Research 35 (19): e132–e132. DOI:10.1093/nar/gkm830
- ↑ M.Y. Jung, L. Lorenz, J.D. Richter, 2006, Translational Control by Neuroguidin, a Eukaryotic Initiation Factor 4E and CPEB Binding Protein, Molecular and Cellular Biology, 26, 11, pp 4277–87, DOI:10.1128/MCB.02470-05, PMID 16705177, PMC 1489097
- ↑ Joel D. Richter, 1999, Cytoplasmic Polyadenylation in Development and Beyond, Microbiology and Molecular Biology Reviews, 63, 2, pp 446–56, PMID 10357857, PMC 98972
- ↑ Takayuki Sakurai, Masahiro Sato, Minoru Kimura, 2005, Diverse patterns of poly(A) tail elongation and shortening of murine maternal mRNAs from fully grown oocyte to 2-cell embryo stages, Biochemical and Biophysical Research Communications, 336, 4, pp 1181–9, PMID 16169522, DOI:10.1016/j.bbrc.2005.08.250
- ↑ R. Taft, 2008, Virtues and limitations of the preimplantation mouse embryo as a model system, Theriogenology, 69, 1, pp 10–6, PMID 18023855, PMC 2239213, DOI:10.1016/j.theriogenology.2007.09.032
- ↑ J. Richter, 2007, CPEB: a life in translation , Trends in Biochemical Sciences, 32, 6, pp 279–85, PMID 17481902, DOI:10.1016/j.tibs.2007.04.004
- ↑ Maria Piqué, José Manuel López, Sylvain Foissac, Roderic Guigó, Raúl Méndez, 2008, A Combinatorial Code for CPE-Mediated Translational Control, Cell, 132, 3, PMID 18267074, pp 434–48, DOI:10.1016/j.cell.2007.12.038
- ↑ P. Benoit, C. Papin, J.E. Kwak, M. Wickens, M. Simonelig, 2008, PAP- and GLD-2-type poly(A) polymerases are required sequentially in cytoplasmic polyadenylation and oogenesis in Drosophila, Development, 135, 11, pp 1969–79, PMID 18434412, DOI:10.1242/dev.021444
- ↑ Mary Edmonds, Richard Abrams, Polynucleotide Biosynthesis: Formation of a Sequence of Adenylate Units from Adenosine Triphosphate by an Enzyme from Thymus Nuclei, The Journal of Biological Chemistry, 235, 4, blz. 1142–9, 1960, PMID 13819354
- ↑ D.F. Colgan , J.L. Manley, Mechanism and regulation of mRNA polyadenylation, 1997, Genes & Development, 11, 21, blz. 2755–66, DOI:10.1101/gad.11.21.2755
- ↑ M. Edmonds, 2002, Progress in Nucleic Acid Research and Molecular Biology at chapter A history of poly A sequences: from formation to factors to function at series Progress in Nucleic Acid Research and Molecular Biology, 71, blz. 285–389, DOI:10.1016/S0079-6603(02)71046-5, ISBN 978-0-12-540071-8
- ↑ M. Edmonds, 1971, Polyadenylic Acid Sequences in the Heterogeneous Nuclear RNA and Rapidly-Labeled Polyribosomal RNA of HeLa cells: Possible Evidence for a Precursor Relationship, Proceedings of the National Academy of Sciences, 68, 6 blz.1336–40, DOI:10.1073/pnas.68.6.1336
- ↑ M. Edmonds, 2002, "A history of poly A sequences: from formation to factors to function". Progress in Nucleic Acid Research and Molecular Biology Volume 71. Progress in Nucleic Acid Research and Molecular Biology. 71. pp. 285–389. DOI:10.1016/S0079-6603(02)71046-5. ISBN 978-0-12-540071-8








